คำอธิบาย
คำอธิบายสินค้า
ชุดเตรียมไลบรารี RNA แบบดูอัลโหมด Hieff NGS™ Ultima เป็นชุดสร้างไลบรารีการจัดลำดับ RNA ทั้งหมดสำหรับแพลตฟอร์มการจัดลำดับ Illumina® และ MGI® ซึ่งรวมถึงรีเอเจนต์การแยกส่วนของ RNA รีเอเจนต์การถอดรหัสย้อนกลับ รีเอเจนต์การสังเคราะห์ ds-cDNA แบบธรรมดาและเฉพาะสาย และรีเอเจนต์การขยายไลบรารี ไลบรารีการจัดลำดับสามารถสร้างขึ้นตามด้วยชุดการทำให้บริสุทธิ์ mRNA หรือชุดการกำจัด rRNA โมดูลการสังเคราะห์สองสายมีบัฟเฟอร์สองตัวเพื่อตอบสนองความต้องการไลบรารีแบบธรรมดาหรือไลบรารีเฉพาะสาย ในจำนวนนั้น dTTP จะถูกแทนที่ด้วย dUTP ในบัฟเฟอร์การสังเคราะห์สองสายเฉพาะสาย ดังนั้นจึงสามารถเพิ่ม dUTP ลงในสายที่สองของ cDNA ได้ DNA โพลิเมอเรสที่มีความเที่ยงตรงสูงที่ใช้ในชุดนี้ไม่สามารถขยายเทมเพลต DNA ที่มียูราซิลได้ ทำให้ได้ความเฉพาะเจาะจงของสาย สารเคมีที่จัดเตรียมไว้ทั้งหมดได้ผ่านการควบคุมคุณภาพและการตรวจสอบการทำงานที่เข้มงวด เพื่อให้แน่ใจว่าการสร้างห้องสมุดมีความเสถียรและทำซ้ำได้ในระดับสูงสุด
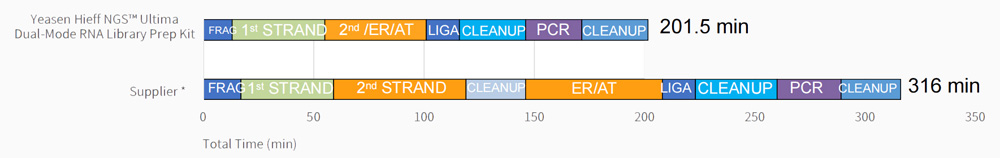
กระบวนการทำงาน
ส่วนประกอบของผลิตภัณฑ์
| ส่วนประกอบ | 12308ES24 | 12308ES96 | |||
| 12308-ก | 2× เฟร็ก/ไพรม์บัฟเฟอร์ | 250 ไมโครลิตร | 930 ไมโครลิตร | ||
| 12308-ข | ส่วนผสมเอนไซม์สายที่ 1 | 48 ไมโครลิตร | 192 ไมโครลิตร | ||
| 12308-ค | รีเอเจนต์ความจำเพาะของสาย | 150 ไมโครลิตร | 580 ไมโครลิตร | ||
| 12308-ง | บัฟเฟอร์สายที่ 2 (dNTP) | 720 ไมโครลิตร | 2×1440 ไมโครลิตร | ||
| 12308-อี | บัฟเฟอร์สายที่ 2 (dUTP) | 720 ไมโครลิตร | 2×1440 ไมโครลิตร | ||
| 12308-ฟ. | 2nd Strand เอนไซม์มาสเตอร์มิกซ์ | 120 ไมโครลิตร | 480 ไมโครลิตร | ||
| 12308-จี | สารเพิ่มประสิทธิภาพการรัด | 720 ไมโครลิตร | 2×1440 ไมโครลิตร | ||
| 12308-ห | ดีเอ็นเอไลเกส T4 แบบใหม่ | 120 ไมโครลิตร | 480 ไมโครลิตร | ||
| 12308-ไอ | 2×ซุปเปอร์คาเนซ® II มิกซ์ไฮไฟ | 600 ไมโครลิตร | 2×1200 ไมโครลิตร | ||
| 12308-ก | นิวคลีเอสฟรี H2O | 300 ไมโครลิตร | 1,000 ไมโครลิตร | ||
หมายเหตุ: ชุดนี้ใช้งานได้กับทั้งแพลตฟอร์ม Illumina และ MGI แต่มี Illumina เพิ่มเติม หรือ เอ็มจีไอ ไพรเมอร์มิกซ์ (Cat# 13335 ไพรเมอร์มิกซ์สำหรับ Illumina และไพรเมอร์มิกซ์ Cat# 13334 สำหรับ MGI ) เป็น ที่จำเป็น.
การขนส่งและการเก็บรักษา
ส่วนประกอบชุดเตรียมไลบรารี mRNA แบบดูอัลโหมด Hieff NGS™ Ultima ในกล่อง I จะจัดส่งพร้อมกับถุงน้ำแข็ง และสามารถจัดเก็บได้ที่อุณหภูมิ 2-8°C เป็นเวลาหนึ่งปี
ส่วนประกอบชุดเตรียมไลบรารี mRNA แบบดูอัลโหมด Hieff NGS™ Ultima ในกล่อง II จะจัดส่งพร้อมน้ำแข็งแห้งและสามารถจัดเก็บที่อุณหภูมิ -20°C ได้นานหนึ่งปี
ข้อควรระวัง
1 การดำเนินการ
1.1 เพื่อความปลอดภัยและสุขภาพของคุณ โปรดสวมอุปกรณ์ป้องกันส่วนบุคคล (PPE) เช่น เสื้อคลุมห้องปฏิบัติการและถุงมือแบบใช้แล้วทิ้ง เมื่อใช้งานผลิตภัณฑ์นี้ ผลิตภัณฑ์นี้มีไว้สำหรับใช้ในการวิจัยเท่านั้น!
1.2 ละลายส่วนผสมที่อุณหภูมิห้อง ผสมให้เข้ากันโดยพลิกขึ้นลงหลายๆ ครั้ง ปั่นให้ละเอียดสักครู่แล้ววางบนน้ำแข็งเพื่อใช้งาน
1.3 ขอแนะนำให้ทำปฏิกิริยาแต่ละขั้นตอนในเครื่องปั่นความร้อนที่มีฝาปิดที่อุ่นไว้ ควรอุ่นเครื่องปั่นความร้อนจนถึงอุณหภูมิที่ตั้งไว้ก่อนใช้งาน
1.4 จำเป็นต้องมีอุปกรณ์ที่ปราศจากการปนเปื้อน RNase และทำความสะอาดพื้นที่ทดลองเป็นประจำ
1.5 การดำเนินการที่ไม่เหมาะสมอาจทำให้เกิดการปนเปื้อนของละอองลอย ซึ่งส่งผลต่อความแม่นยำของผลลัพธ์ แนะนำให้แยกบริเวณการผสมปฏิกิริยา PCR และบริเวณการทดสอบการทำให้บริสุทธิ์ของผลิตภัณฑ์ PCR ออกจากกันทางกายภาพโดยบังคับ ติดตั้งอุปกรณ์ เช่น ปิเปตเฉพาะทางสำหรับการสร้างห้องสมุด
2 อะแดปเตอร์เชื่อมต่อ
2.1 ชุด Illumina หรือ MGI Long Adapter (Barcoded Adapter) และชุด Short Adapter มีให้เลือกใช้ตามความต้องการในการทดลองของพวกเขา
2.2 ขอแนะนำให้เลือกใช้อะแดปเตอร์เชิงพาณิชย์ที่มีคุณภาพสูง หากเลือกอะแดปเตอร์ที่ผลิตเอง โปรดฝากบริษัทที่มีประสบการณ์ในการสังเคราะห์ไพรเมอร์ NGS และแสดงความคิดเห็นเกี่ยวกับความจำเป็นในการควบคุมการปนเปื้อนอย่างเข้มงวด นอกจากนี้ ขอแนะนำให้เตรียมสารละลายแอนนีลลิ่ง DNA ในม้านั่งที่สะอาด และใช้อะแดปเตอร์เพียงประเภทเดียวในแต่ละครั้งเพื่อป้องกันการปนเปื้อนข้าม
2.3 โปรดละลายอะแดปเตอร์บนน้ำแข็งหรือที่อุณหภูมิ 4°C เมื่อใช้งานที่อุณหภูมิห้อง อุณหภูมิในห้องปฏิบัติการไม่ควรเกิน 25°C เพื่อป้องกันไม่ให้อะแดปเตอร์เสียสภาพ
2.4 ความเข้มข้นของอะแดปเตอร์ส่งผลโดยตรงต่อประสิทธิภาพการรัดและผลผลิตของคลังข้อมูล ปริมาตรของอะแดปเตอร์ที่เพิ่มเข้าไปในชุดอุปกรณ์จะถูกกำหนดไว้ที่ 5 μl ขอแนะนำให้เจือจางอะแดปเตอร์ด้วยบัฟเฟอร์ 0.1×TE และอะแดปเตอร์ที่เจือจางสามารถเก็บไว้ที่อุณหภูมิ 4°C เป็นเวลา 48 ชั่วโมง ตารางที่ 1 แสดงจำนวนอะแดปเตอร์ที่แนะนำสำหรับปริมาณ RNA อินพุตที่แตกต่างกัน
ตาราง 1-1 ปริมาณอะแดปเตอร์ Illumina ที่แนะนำสำหรับ RNA อินพุตที่แตกต่างกัน
| อินพุต RNA ทั้งหมด | อิลลูมิน่า ความเข้มข้นของสต็อกอะแดปเตอร์ |
| 10 ง | 1 ไมโครโมลาร์ |
| 100 นาโนกรัม | 1.5 ไมโครโมลาร์ |
| 500 นาโนกรัม | 3 ไมโครโมลาร์ |
| ≥1 ไมโครกรัม | 5 ไมโครโมลาร์ |
ตาราง 1-2 ปริมาณอะแดปเตอร์ MGI® ที่แนะนำสำหรับ RNA อินพุตที่แตกต่างกัน
| อินพุต RNA ทั้งหมด | เอ็มจีไอ ความเข้มข้นของสต็อกอะแดปเตอร์ |
| 100-499 ง | 2 ไมโครโมลาร์ |
| 500-4000 นาโนกรัม | 5 ไมโครโมลาร์ |
*การใช้งานอะแดปเตอร์สามารถปรับเปลี่ยนได้ตามประเภทของตัวอย่าง RNA ทั้งหมดและปริมาณอินพุตที่แตกต่างกัน
3 การขยายห้องสมุด
3.1 บนพื้นฐานของดีเอ็นเอโพลีเมอเรสรุ่นแรก ดีเอ็นเอโพลีเมอเรสที่มีความเที่ยงตรงสูงในชุดทดสอบได้ปรับปรุงความสม่ำเสมอของการขยายสัญญาณให้ดีขึ้นอย่างมาก และไม่มีความลำเอียงในการขยายสัญญาณเลย
3.2 หากเชื่อมต่ออะแดปเตอร์ดัชนี (เรียกอีกอย่างว่าอะแดปเตอร์ยาวหรืออะแดปเตอร์ Y ขนาดใหญ่) เข้ากับ DNA เป้าหมาย สามารถใช้ไพรเมอร์ผสมที่มีให้ในชุดนี้เพื่อการขยายได้ หากใช้ "อะแดปเตอร์สั้น" หรือ "อะแดปเตอร์ Y ขนาดเล็ก" สำหรับการเชื่อมต่อ DNA จำเป็นต้องใช้ไพรเมอร์ดัชนีเพื่อการขยาย
3.3 ควรควบคุมจำนวนรอบการขยายอย่างเคร่งครัด การขยายที่ไม่เพียงพออาจส่งผลให้ผลผลิตของไลบรารีต่ำ การขยายมากเกินไปอาจทำให้เกิดอคติที่เพิ่มขึ้น ข้อผิดพลาด การอ่านซ้ำ ผลิตภัณฑ์ไคเมอริก และการกลายพันธุ์แบบขยายสะสม ตารางที่ 2 แสดงจำนวนรอบที่แนะนำสำหรับการขยาย PCR
ตารางที่ 2 จำนวนรอบที่แนะนำในการสร้างคลัง RNA*
| อินพุต RNA ทั้งหมด | จำนวนรอบ | |
| ไม่ติดขัด | ติดแหงกอยู่ | |
| 10 ง | 15 | 15 |
| 100 นาโนกรัม | 14 | 14 |
| 500 นาโนกรัม | 12 | 13 |
| 1 ไมโครกรัม | 11 | 12 |
หมายเหตุ: * ผลผลิตของห้องสมุดไม่ได้ขึ้นอยู่กับปริมาณอินพุตและจำนวนรอบการขยายเท่านั้น แต่ยังได้รับผลกระทบจากคุณภาพของตัวอย่าง สภาวะการแยกส่วน และเงื่อนไขการคัดแยก ในกระบวนการสร้างห้องสมุด ให้เลือกเงื่อนไขที่เหมาะสมที่สุดตามสถานการณ์จริง
การทำความสะอาด DNA ตามลูกปัด 4 แบบและการเลือกขนาด
4.1 มีหลายขั้นตอนในกระบวนการสร้างห้องสมุดที่ต้องใช้ลูกปัดแม่เหล็กสำหรับฟอก DNA เราขอแนะนำลูกปัดคัดเลือก DNA Hieff NGS™ (
4.2 ควรปรับสมดุลลูกปัดแม่เหล็กที่อุณหภูมิห้องก่อนใช้งาน มิฉะนั้น ผลผลิตจะลดลง และส่งผลต่อการเลือกขนาด
4.3 ควรผสมลูกปัดแม่เหล็กให้เข้ากันโดยใช้วิธีวอร์เท็กซ์หรือปิเปตก่อนใช้งาน
4.4 ห้ามดูดลูกปัดออกเมื่อถ่ายโอนของเหลวส่วนบน แม้ว่าจะมีลูกปัดเพียงเล็กน้อยก็อาจส่งผลต่อปฏิกิริยาต่อไปนี้ได้
4.5 ควรเตรียมเอธานอล 80% ใหม่ๆ มิฉะนั้น จะส่งผลกระทบต่อประสิทธิภาพการกู้คืน
4.6 ควรทำให้ลูกปัดแม่เหล็กแห้งที่อุณหภูมิห้องก่อนทำการชะผลิตภัณฑ์ออก ความแห้งที่ไม่เพียงพอจะทำให้เอธานอลที่เหลือส่งผลต่อปฏิกิริยาที่เกิดขึ้นในภายหลังได้ง่าย ความแห้งที่มากเกินไปจะทำให้ลูกปัดแม่เหล็กแตกร้าวและลดผลผลิตในการทำให้บริสุทธิ์ โดยปกติแล้ว การทำให้แห้งที่อุณหภูมิห้องเป็นเวลา 3-5 นาทีก็เพียงพอที่จะทำให้ลูกปัดแห้งสนิท
4.7 หากจำเป็น ตัวอย่าง DNA ที่ผ่านการทำให้บริสุทธิ์หรือเลือกขนาดแล้วซึ่งแยกออกจากบัฟเฟอร์ TE สามารถเก็บไว้ที่อุณหภูมิ 4°C เป็นเวลา 1-2 สัปดาห์หรือที่อุณหภูมิ -20°C เป็นเวลาหนึ่งเดือน
5 การวิเคราะห์คุณภาพห้องสมุด
5.โดยปกติแล้ว คุณภาพของห้องสมุดที่สร้างขึ้นสามารถประเมินได้โดยการกระจายความยาวและการตรวจจับความเข้มข้น
5.2 การตรวจจับความเข้มข้นของห้องสมุด: วิธีการที่ใช้สีย้อมเรืองแสง DNA แบบสองสาย เช่น Qubit®, PicoGreen® เป็นต้น การวัดปริมาณแบบสัมบูรณ์โดยใช้ qPCR
5.3 วิธีการที่ใช้การตรวจจับแบบสเปกตรัม เช่น NanoDrop® เป็นต้น ไม่สามารถนำไปใช้กับการตรวจจับความเข้มข้นของห้องสมุดได้
5.4 แนะนำให้ใช้ qPCR เพื่อตรวจหาความเข้มข้นของไลบรารี: ผ่าน Qubit® PicoGreen® และวิธีการอื่นๆ ที่ใช้สีย้อมเรืองแสงดีเอ็นเอแบบสองสาย ทำให้ไม่สามารถแยกแยะระหว่างผลิตภัณฑ์ที่เชื่อมต่อกับอะแดปเตอร์ที่ปลายด้านหนึ่ง ผลิตภัณฑ์ที่ไม่ได้เชื่อมต่อกับอะแดปเตอร์ที่ปลายทั้งสองด้าน และผลิตภัณฑ์สายคู่ที่ไม่สมบูรณ์อื่นๆ ได้อย่างมีประสิทธิภาพ การวัดปริมาณ qPCR แบบสัมบูรณ์นั้นใช้หลักการของการขยาย PCR ซึ่งจะวัดปริมาณไลบรารีทั้งหมดของอะแดปเตอร์ที่ปลายทั้งสองด้านของตัวอย่างเท่านั้น (ไลบรารีที่สามารถเรียงลำดับได้) โดยไม่รวมถึงการรบกวนของไลบรารีที่ไม่เรียงลำดับซึ่งไม่ได้เชื่อมต่อกับอะแดปเตอร์ที่ปลายด้านเดียวหรือปลายสองด้าน
5.5 การตรวจจับการกระจายความยาวห้องสมุดสามารถทำได้โดยใช้ Agilent Bioanalyzer 2100 และอุปกรณ์อื่นๆ โดยใช้หลักการของอิเล็กโทรโฟรีซิสแบบแคปิลลารีหรือไมโครฟลูอิดิกส์
เอกสาร:
12308ES-Hieff NGS™ Ultima Dual-mode RNA Library Prep Kit-Ver.EN20230327.pdf
การอ้างอิง & เอกสารอ้างอิง:
[1] การถอดรหัสไมโครไบโอมในลำไส้ของปลาคาร์ปโดยใช้แนวทางมัลติโอมิกส์
เอ็ม ลี่, เอช เหลียง, เอช หยาง, คิว ติง, อาร์ เซี่ย, เจ เฉิน, ดับเบิลยู โจว… - ไมโครไบโอม, 2024 IF:19.4
[2] การคัดกรอง CRISPR แบบรวมระบุว่า P-Bodies เป็นตัวระงับการเปลี่ยนผ่านจากเยื่อบุผิวเป็นเนื้อเยื่อเกี่ยวพันของมะเร็ง
L Fang, L Zhang, M Wang, Y He, J Yang, Z Huang… - การวิจัยโรคมะเร็ง, 2024 IF:12.7
[3] เปปไทด์ 1 ที่ได้จาก Klotho ยับยั้งการแก่ของเซลล์ในไตที่เป็นไฟโบรซิสโดยฟื้นฟูการแสดงออกของ Klotho ผ่านการควบคุมหลังการถอดรหัส
เอ็กซ์ จาง, แอล ลี่, เอช แทน, เอ็กซ์ ฮ่อง, คิว หยวน, เอฟเอฟ โฮ… - Theranostics, 2024 IF:12.4
[4] เซลล์ต้นกำเนิดเคลือบบางส่วนจากโครงกระดูกภายนอกเพื่อการฟื้นฟูกล้ามเนื้อหัวใจที่ขาดเลือด
H He, Y Yuan, Y Wu, J Lu, X Yang, K Lu… - วัสดุขั้นสูง, 2023 IF: 29.4
[5] นวัตกรรมจีโนมและการเชื่อมโยงการควบคุมใหม่ระหว่างวิวัฒนาการของสกุลฝ้าย Gossypium
เอ็ม หวาง, เจ ลี่, ซี ฉี, วาย หลง, แอล เป่ย, เอ็กซ์ ฮวง… - Nature Genetics, 2022 IF:41.379
[6] อัลลีลความเสี่ยง MTMR3 ช่วยเพิ่มภูมิคุ้มกัน IgA ที่เกิดจาก Toll Like Receptor 9 ในโรคไตจาก IgA
Y Wang, T Gan, S Qu, L Xu, Y Hu, L Liu, S Shi, J Lv… - Kidney International, 2023 IF:19.6
[7] แบ่งการเสริมของบรรณาธิการฐานเพื่อลดการแก้ไขที่ไม่ตรงเป้าหมาย
X Xiong, K Liu, Z Li, FN Xia, XM Ruan, X He, JF Li - พืชธรรมชาติ 2023 IF:18.6
[8] การออกแบบเวสิเคิลเยื่อหุ้มภายนอกของแบคทีเรียที่ห่อหุ้มอะดีโนไวรัสที่ทำลายเซลล์มะเร็งช่วยเพิ่มประสิทธิภาพของการบำบัดด้วยไวรัสสำหรับมะเร็งโดยเพิ่มการทำงานของออโตฟาจีของเซลล์เนื้องอก
ดับเบิลยู บาน, เอ็ม ซัน, เอช ฮวง, ดับเบิลยู ฮวง, เอส แพน… - Nature Communications, 2023 IF:16.6
[9] ความต้านทานของเซลล์มะเร็งต่อ IFNγ สามารถเกิดขึ้นได้จากกิจกรรมเส้นทางการซ่อมแซม Double-Strand Break ที่ได้รับการปรับปรุง
T Han, X Wang, S Shi, W Zhang, J Wang, Q Wu… - การวิจัยภูมิคุ้มกันวิทยามะเร็ง, 2023 IF: 12.0
[10] การบำบัดแบบผสมผสานโดยใช้ระบบส่งมอบนาโนเลียนแบบชีวภาพเป้าหมายคู่เพื่อเอาชนะความต้านทานซิสแพลตินในมะเร็งเซลล์ตับ
Y Huang, Q Kou, Y Su, L Lu, X Li, H Jiang… - วารสารนาโนเทคโนโลยีชีวภาพ, 2023 IF: 10.9
[11] PIAS3 ส่งเสริมการเกิดเฟอร์โรพโทซิสโดยควบคุม TXNIP ผ่านทางเส้นทางการส่งสัญญาณ TGF-β ในมะเร็งเซลล์ตับ
W Bao, J Wang, K Fan, Y Gao, J Chen - การวิจัยด้านเภสัชวิทยา 2023 IF:9.3
[12] การระบุเป้าหมายโปรตีนที่จับกับ RNA ด้วย HyperTRIBE ใน Saccharomyces cerevisiae
W Piao, C Li, P Sun, M Yang, Y Ding, W Song… - วารสารนานาชาติด้านวิทยาศาสตร์โมเลกุล 2023 IF:5.6
[13] จุลินทรีย์ควบคุมการเปลี่ยนแปลงของวงจรชีวิตและพลวัตของเซลล์นีมาโตไซต์ในแมงกะพรุน
เอส เป็ง, แอล เย, วาย ลี่, เอฟ หวัง, ที ซุน, แอล หวัง, ดับเบิลยู เฮา… - Iscience, 2023 IF: 5.08
[14] โปรไฟล์ทรานสคริปโตมและไมโครอาร์เอ็นเอเผยให้เห็นเครือข่ายควบคุมและตัวควบคุมหลักของการก่อตัวของไซเลมรองในป็อปลาร์ “84K”
H Wang, P Zhao, Y He, Y Su, X Zhou… - วารสารวิทยาศาสตร์โมเลกุลนานาชาติ, 2023 IF:5.6
การชำระเงินและความปลอดภัย
ข้อมูลการชำระเงินของคุณได้รับการดำเนินการอย่างปลอดภัย เราไม่เก็บรายละเอียดบัตรเครดิตและไม่สามารถเข้าถึงข้อมูลบัตรเครดิตของคุณได้
การสอบถาม
คุณอาจชอบ
คำถามที่พบบ่อย
ผลิตภัณฑ์นี้มีวัตถุประสงค์เพื่อการวิจัยเท่านั้น และไม่ได้มีวัตถุประสงค์เพื่อใช้ในการรักษาหรือวินิจฉัยโรคในมนุษย์หรือสัตว์ ผลิตภัณฑ์และเนื้อหาได้รับการคุ้มครองโดยสิทธิบัตร เครื่องหมายการค้า และลิขสิทธิ์ที่เป็นของ
แอปพลิเคชั่นบางตัวอาจต้องใช้สิทธิ์ในทรัพย์สินทางปัญญาของบุคคลที่สามเพิ่มเติม

