จะเลือกอะแดปเตอร์ NGS ของคุณอย่างไร?
อะแดปเตอร์ NGS ซึ่งเป็นส่วนสำคัญของไลบรารีการจัดลำดับยีนรุ่นถัดไป มีบทบาทในการเชื่อมต่อชิ้นส่วน DNA ที่ผ่านการทดสอบกับเซลล์โฟลว์ (ชิปการจัดลำดับยีน) ประสิทธิภาพของข้อต่อเป็นปัจจัยสำคัญในการกำหนดคุณภาพและผลผลิตของไลบรารี อะแดปเตอร์ NGS คืออะไร อะแดปเตอร์ NGS มีประเภททั่วไปอะไรบ้าง และจะเลือกอะแดปเตอร์ NGS ที่เหมาะสมที่สุดสำหรับแพลตฟอร์มการจัดลำดับยีนของคุณได้อย่างไร
1. NGS Adapter คืออะไร?
2. ปัจจัยใดบ้างที่คุณควรพิจารณาเมื่อเลือกดัชนี NGS?
3. ประเภทดัชนีทั่วไปมีอะไรบ้าง?
4. อะแดปเตอร์ NGS มีประเภททั่วไปอะไรบ้าง?
- อะแดปเตอร์ UMI
- อะแดปเตอร์ครบชุด
- อะแดปเตอร์ไม่สมบูรณ์
- อะแดปเตอร์ Tn5
5. จะเลือกอะแดปเตอร์ NGS ที่เหมาะสมสำหรับแพลตฟอร์มการจัดลำดับของคุณได้อย่างไร
6. เรื่องการอ่าน
1. NGS Adapter คืออะไร?
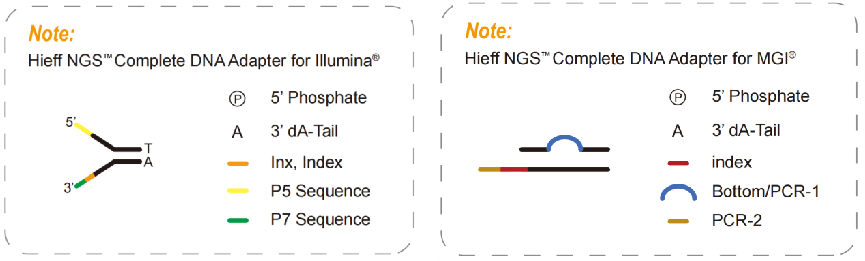
อะแดปเตอร์ NGS เป็นชุดอะแดปเตอร์ในการจัดลำดับนิวคลีโอไทด์สั้นที่มีลำดับที่ทราบแล้ว โดยจะเชื่อมต่อกับปลายทั้งสองด้านของชิ้นส่วนกรดนิวคลีอิกเป้าหมาย ในระหว่างการจัดลำดับ อะแดปเตอร์จะเริ่มจัดลำดับโดยไฮบริดไดซ์กับลำดับที่ทราบแล้วบนเซลล์โฟลว์เพื่อรวมไลบรารีเข้ากับชิป แล้วโครงสร้างของอะแดปเตอร์ NGS คืออะไร?
โดยใช้แพลตฟอร์ม Illumina เป็นตัวอย่าง อะแดปเตอร์ NGS สามารถแบ่งออกได้เป็น 3 ส่วน:
P5 และ P7: ลำดับที่รวมกับปลาย P5 และ P7 บนเซลล์โฟลว์เพื่อแก้ไขไลบรารีบนชิปการจัดลำดับ อำนวยความสะดวกในการตอบสนองคลัสเตอร์ผ่าน Bridge-PCR
Rd1 SP และ Rd2 SP (ไพรเมอร์ลำดับ Read1/Read2) - บริเวณที่จับกับไพรเมอร์ลำดับ ระบุตำแหน่งที่ลำดับเริ่มถูกอ่าน
ดัชนี (เรียกอีกอย่างว่าบาร์โค้ด): ลำดับสังเคราะห์ที่รู้จักซึ่งใช้เพื่อแยกแยะตัวอย่างต่างๆ ในการจัดลำดับของไลบรารีแบบผสม
มะเดื่อ. 1 ไลบรารีดัชนีเดียวของแพลตฟอร์ม Illumina

มะเดื่อ. 2 ไลบรารีดัชนีปลายเดียวของแพลตฟอร์ม MGI
เมื่อปริมาณงานในการเรียงลำดับเพิ่มมากขึ้น ก็สามารถเรียงลำดับตัวอย่างได้หลายตัวอย่างในเวลาเดียวกัน ดังนั้น การแยกแยะตัวอย่างที่หลากหลายจึงมีความสำคัญเป็นอย่างยิ่ง ดังที่ได้กล่าวไปก่อนหน้านี้ ลำดับดัชนีของอะแดปเตอร์ NGS ใช้เพื่อแยกแยะตัวอย่างต่างๆ ในการจัดลำดับรุ่นถัดไป (NGS) คุณควรพิจารณาปัจจัยใดบ้างเมื่อเลือกดัชนี โปรดอ่านต่อ...
2. ปัจจัยใดบ้างที่คุณควรพิจารณาเมื่อเลือกดัชนี?
ดัชนีโดยทั่วไปจะมีความยาว 6nt-18nt และแบ่งออกเป็นดัชนีเดี่ยวและดัชนีคู่ตามจำนวนดัชนี ดัชนีคู่จะอยู่ที่ปลายทั้งสองด้านของชิ้นส่วนที่จะทดสอบ ควรพิจารณาความสมดุลของฐานและความสมดุลของฟลูออเรสเซนต์เมื่อเลือกชุดดัชนี
ยอดคงเหลือฐานหมายถึงยอดคงเหลือระหว่างดัชนีหลายตัว แทนที่จะเป็นยอดคงเหลือฐานในดัชนีตัวเดียว จำเป็นต้องพิจารณาจากทั้งประเภทฐานและการกระจายฐาน หลักการรวมกันคือต้องรวมฐานทั้งสี่ A/T/C/G ในกลุ่มดัชนีเดียวกัน และสัดส่วนของฐานทั้งสี่นี้จะใกล้เคียงกัน คิดเป็นประมาณ 25% ตามลำดับ
ความสมดุลของสัญญาณฟลูออเรสเซนต์หมายถึงการเลือกเพื่อให้แน่ใจว่าสัญญาณฟลูออเรสเซนต์มีความสมดุลเมื่อไม่สามารถรับประกันความสมดุลของฐานได้ ในเครื่องเรียงลำดับ 4 ช่องสัญญาณในแพลตฟอร์ม Illumina นั้น dG/dT จะถูกติดฉลากด้วยฟลูออเรสเซนต์สีเขียว และ dC/dA จะถูกติดฉลากด้วยฟลูออเรสเซนต์สีแดง ในระหว่างการเรียงลำดับ จะต้องมีสัญญาณเรืองแสงทั้งสีเขียวและสีแดงในแต่ละรอบเพื่อให้แน่ใจว่าการเรียงลำดับจะประสบความสำเร็จ ดังนั้นควรพิจารณาความสมดุลระหว่างสัญญาณสีเขียวและสัญญาณสีแดงเมื่อทำการเลือกดัชนี
3. ประเภทดัชนีทั่วไปมีอะไรบ้าง?
ดัชนีคู่ทั่วไปมักได้แก่ ดัชนีคู่ที่ไม่ซ้ำใคร (UDI), บาร์โค้ดคู่ที่ไม่ซ้ำใคร (UDB) และดัชนีคู่แบบรวม (CDI) ซึ่งช่วยลดการกระโดดดัชนีและการกำหนดที่ไม่ถูกต้องได้อย่างมาก
UDI&UDB: ดัชนีทั้งสองด้านเป็นแบบหนึ่งต่อหนึ่ง ออกแบบเป็นกลุ่ม และสามารถตรวจสอบไขว้กันได้ทั้งสองด้าน
ชุดไพรเมอร์ Stubby UDI สำหรับ Illumina จัดทำโดย
CDI: ดัชนีทั้งสองด้านสามารถรวมกันได้ตามข้อกำหนดบางประการเพื่อสร้างไลบรารีดัชนีสองด้าน
ไพรเมอร์ A 384 CDI สำหรับ Illumina ชุดที่ 1-ชุดที่ 2 จัดทำโดย
เพื่อปรับปรุงประสิทธิภาพผลผลิตและการขยายสัญญาณ และลดต้นทุนการจัดลำดับ Illumina จึงได้แนะนำเซลล์การไหลของอาร์เรย์ (PFCT) และเทคโนโลยีคลัสเตอร์การขยายสัญญาณเฉพาะ (ExAmp) สำหรับ Novaseq และเครื่องจัดลำดับผลผลิตสูงอื่นๆ แต่ได้ขยายปรากฏการณ์ความไม่ตรงกันของฉลากตัวอย่างและการกระโดดดัชนีโดยไม่ได้ตั้งใจ

รูปที่ 3 แบบจำลองเครื่องมือ Illumina ที่แตกต่างกันใช้เซลล์การไหลแบบไม่มีรูปแบบหรือเซลล์การไหลแบบมีรูปแบบ
เพื่อชดเชยปัญหาการวนซ้ำของดัชนีที่เน้นโดยแพลตฟอร์มการจัดลำดับเช่น HiSeq3000/4000, HiSeq X Series และ NovaSeq บริษัท Illumina เสนอแนวทางการวางดัชนีไว้ที่ปลายทั้งสองด้านของไลบรารี ซึ่งสามารถทำการตรวจสอบแบบทวิภาคีและกำจัดอะแดปเตอร์ที่ไม่ตรงกันได้ เมื่อใช้ดัชนีที่ไม่ซ้ำกันที่ปลายทั้งสองด้าน อัตราการจัดสรรข้อผิดพลาดของดัชนีจะลดลงเหลือ 0.01% เมื่อเปรียบเทียบกับวิธีการรวมกลุ่มการเรียงสับเปลี่ยนดัชนีแบบเดิม การวนซ้ำของดัชนีจะลดลงสองลำดับความสำคัญ
ในการสร้างไลบรารีที่ปราศจาก PCR จะมีอะแดปเตอร์ดัชนีปลายเดียวให้เลือกใช้ ความไม่ตรงกันของฉลากส่วนใหญ่มักเกิดจากข้อผิดพลาดในการเรียงลำดับ โดยรวมแล้ว อัตราความไม่ตรงกันของฉลากนั้นต่ำ (เฉลี่ย 0.0004% สูงสุด 0.001%) อย่างไรก็ตาม ในการสร้างไลบรารีการจับข้อมูลแบบกำหนดเป้าหมาย ปัญหาครอสทอล์คจะทวีความรุนแรงขึ้น เนื่องจากขั้นตอนหลายขั้นตอนจะนำไปสู่ความไม่ตรงกันของฉลาก และโดยปกติแล้วจะใช้อะแดปเตอร์ UDI/UDB/CDI
4.อะแดปเตอร์ NGS มีประเภททั่วไปอะไรบ้าง?
ด้วยการพัฒนาของเทคโนโลยีการจัดลำดับ จึงมีอะแดปเตอร์ประเภทต่างๆ เพิ่มมากขึ้นเรื่อยๆ เช่น อะแดปเตอร์ดัชนีเดี่ยว/ดัชนีคู่ (ดังที่กล่าวถึงในหัวข้อที่ 3) อะแดปเตอร์ UMI อะแดปเตอร์ทรานสโพเซส อะแดปเตอร์สมบูรณ์/ไม่สมบูรณ์ เป็นต้น ซึ่งเหมาะสำหรับสถานการณ์การใช้งานที่หลากหลาย ส่วนนี้จะจัดเรียงอะแดปเตอร์เหล่านี้อย่างเป็นระบบเพื่อให้คุณมีพื้นฐานในการเลือกอะแดปเตอร์
4.1 อะแดปเตอร์ UMI
อะแดปเตอร์ระบุโมเลกุลเฉพาะ (UMI) เป็นเครื่องมือขั้นสูงสำหรับการตรวจจับการกลายพันธุ์ความถี่ต่ำและการวัดปริมาณแบบสัมบูรณ์ UMI เป็นลำดับสังเคราะห์แบบสุ่มที่มีลำดับที่ทราบ สามารถออกแบบเป็นโซ่นิวคลีโอไทด์แบบสุ่มอย่างสมบูรณ์ โซ่นิวคลีโอไทด์ที่เสื่อมสภาพบางส่วน หรือโซ่นิวคลีโอไทด์คงที่ความยาวโดยทั่วไปคือ 10nt (UMI ปลายเดียว) หรือ 5-8nt (UMI ปลายคู่) หน้าที่ของมันคือตรึงสถานะของชิ้นส่วน DNA ก่อนการขยาย และโมเลกุล DNA แต่ละโมเลกุลจะสอดคล้องกับ UMI ดังนั้นในระหว่างการวิเคราะห์ชีวสารสนเทศ มันสามารถแยกแยะเทมเพลต DNA จากแหล่งต่างๆ แยกแยะว่าอันไหนคือการกลายพันธุ์ที่เป็นบวกเท็จซึ่งเกิดจากข้อผิดพลาดแบบสุ่มในกระบวนการขยายและการจัดลำดับด้วย PCR และอันไหนที่ผู้ป่วยเป็นพาหะ เพื่อกรองสัญญาณรบกวนพื้นหลัง ตรวจจับการกลายพันธุ์ความถี่ต่ำและความถี่ต่ำมากได้อย่างแม่นยำ และวัดปริมาณโมเลกุล DNA ที่แตกต่างกันได้แน่นอน มันถูกนำไปใช้กันอย่างแพร่หลายในการตรวจจับการกลายพันธุ์ความถี่ต่ำ โดยเฉพาะอย่างยิ่งในสาขาการวิจัยเนื้องอก

รูปที่ 4 แผนผังของอะแดปเตอร์ UMI โครงสร้างของแพลตฟอร์ม Illumina
4.2 อะแดปเตอร์ครบชุด
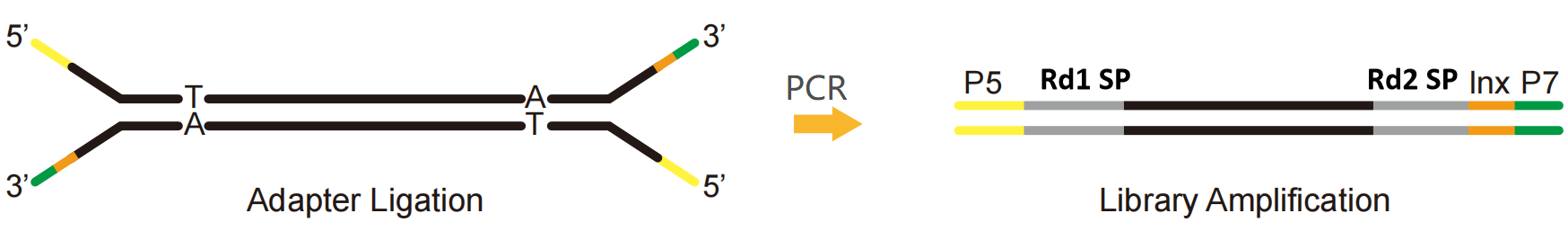
อะแดปเตอร์ครบชุด ซึ่งเป็นผลิตภัณฑ์ที่จำเป็นสำหรับไลบรารีที่ปราศจาก PCR ประกอบด้วยลำดับทั้งหมดที่จำเป็นสำหรับการจัดลำดับ เช่น P5, P7, RdS1 และ RdS2 ในแพลตฟอร์ม Illumina นอกจากนี้ยังมีลำดับดัชนีและลำดับ UMI ตามข้อกำหนดสำหรับการจัดลำดับ ด้วยอะแดปเตอร์ครบชุด จึงสามารถจัดลำดับได้โดยตรงโดยไม่ต้องใส่อะแดปเตอร์อื่นผ่าน PCR ดังนั้น อะแดปเตอร์ครบชุดจึงสามารถใช้สร้างไลบรารีที่ปราศจาก PCR ได้ ไลบรารีที่ปราศจาก PCR สามารถลดอคติการขยาย PCR อัตราข้อผิดพลาด และการทำซ้ำของลำดับ ทำให้ครอบคลุมพื้นที่ GC สูงหรือ AT สูงบางส่วนได้มากขึ้น ซึ่งใช้กันอย่างแพร่หลายในการวิจัยจีโนมประชากร
ผลิตภัณฑ์อะแดปเตอร์ที่สมบูรณ์สำหรับแพลตฟอร์ม Illumina ที่จัดหาโดย
ผลิตภัณฑ์อะแดปเตอร์ที่สมบูรณ์สำหรับแพลตฟอร์ม MGI ที่จัดหาโดย
รูปที่ 5 แผนผังอะแดปเตอร์แบบสมบูรณ์
4.3 อะแดปเตอร์ที่ไม่สมบูรณ์
อะแดปเตอร์ที่ไม่สมบูรณ์จำเป็นต้องแนะนำลำดับอื่น ๆ โดย PCR หลังจากการเชื่อมต่ออะแดปเตอร์เพื่อสร้างอะแดปเตอร์ที่สมบูรณ์ อะแดปเตอร์เหล่านี้มีลักษณะเฉพาะคือประสิทธิภาพการเชื่อมต่อสูงและอัตราไลบรารีที่มีประสิทธิผลสูง กระบวนการ PCR เป็นเอฟเฟกต์การเสริมประสิทธิภาพสำหรับไลบรารีที่สมบูรณ์เพื่อให้แน่ใจว่าไลบรารีที่มีประสิทธิผลมีความเข้มข้น และยังสามารถแนะนำดัชนีปลายคู่และลำดับ UMI ได้อีกด้วย
4.4 อะแดปเตอร์ Tn5
อะแดปเตอร์ Tn5 เชื่อมต่อส่วนหนึ่งของลำดับอะแดปเตอร์กับปลายทั้งสองข้างของชิ้นส่วน DNA ผ่านกิจกรรมเอนไซม์จำกัดเอ็นโดนิวคลีเอสของ Tn5 อะแดปเตอร์นี้ทำการแบ่งส่วนและผูกอะแดปเตอร์พร้อมกันเพื่อประหยัดเวลาและตัวอย่าง ในที่สุด ลำดับลิงก์เกอร์ ดัชนี UMI และลำดับอื่นๆ ที่เหลือจะถูกนำเข้าสู่ PCR เพื่อสร้างไลบรารีที่สมบูรณ์ ซึ่งสามารถใช้เพื่อสร้างไลบรารี Cut&tag ได้
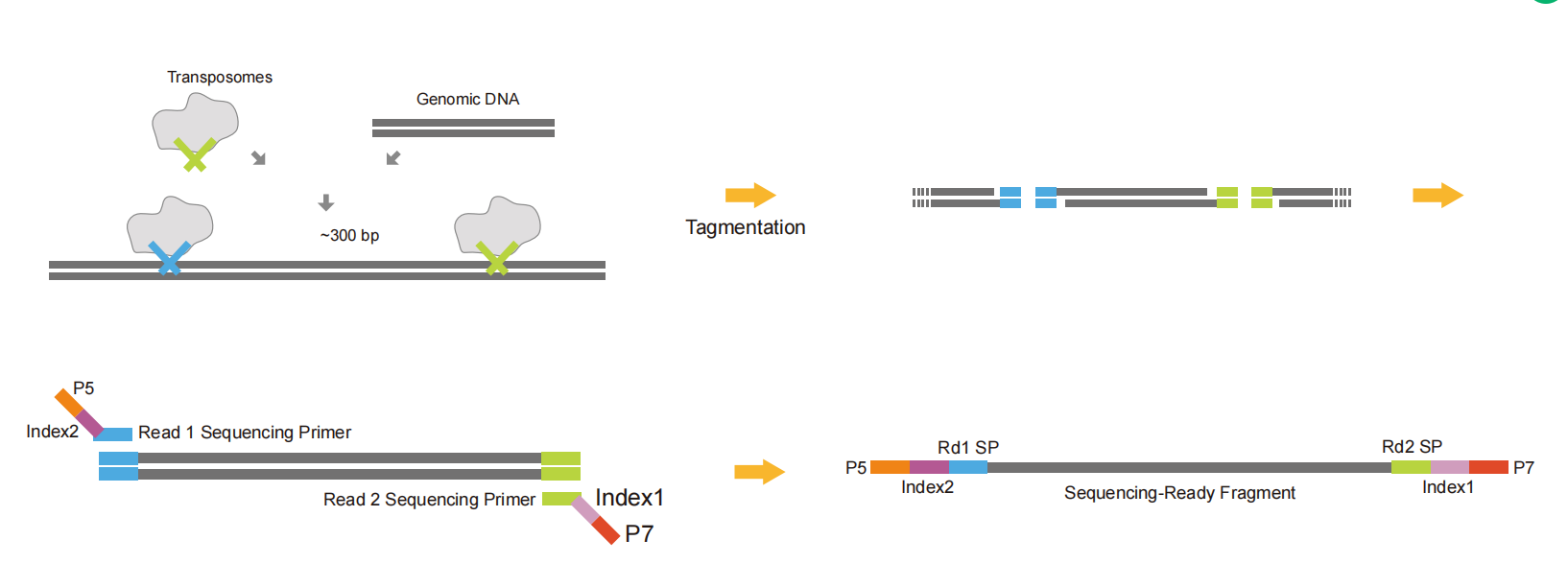
รูปที่ 6 แผนผังของการสร้างไลบรารีอะแดปเตอร์ Tn5
5. จะเลือกอะแดปเตอร์ NGS ที่เหมาะสมสำหรับแพลตฟอร์มการจัดลำดับของคุณได้อย่างไร
ปัจจุบันมีแพลตฟอร์มการจัดลำดับยีนหลักอยู่ 2 แพลตฟอร์ม ได้แก่ Illumina และ MGI
ในแง่ของแพลตฟอร์ม Illumina อะแดปเตอร์ Illumina NGS จัดหาโดย
อะแดปเตอร์ NGS แบบครบชุดและ UDI ไม่ต้องกังวลเรื่องปัญหาการเชื่อมต่อ เหมาะสำหรับลูกค้าที่ต้องการใช้งานง่าย อะแดปเตอร์ NGS ของ CDI มีหลอดน้อยกว่าและมีขนาดเล็ก เหมาะสำหรับลูกค้าที่ต้องการจัดเก็บและพกพาสะดวก ปราศจาก PCR ต้องใช้อะแดปเตอร์ NGS แบบครบชุด
สำหรับ อิลลูมิน่า
| อินทูป | ไฮฟ์ เอ็นจีเอส ดีการเตรียมสอบ NA Lib 384 CDI Primer สำหรับ Illumina ชุดที่ 1 (8*12, ดัชนี 96) | 12412ES |
| ไฮฟ์ เอ็นจีเอส ดีการเตรียมสอบ NA Lib 384 CDI Primer สำหรับ Illumina ชุดที่ 2 (8*12, ดัชนี 96) | 12413ES | |
| ไพรเมอร์ Hieff NGS® RNA Lib Prep 384 CDI สำหรับ Illumina ชุดที่ 1 (ดัชนี 96) | 12414ES | |
| ไพรเมอร์ Hieff NGS® RNA Lib Prep 384 CDI สำหรับ Illumina ชุดที่ 1 (ดัชนี 96) | 12415ES | |
| ในจาน | ชุดไพรเมอร์ Hieff NGS® Stubby UDI สำหรับ Illumina (ดัชนี 1-384) ชุด 1-4 | 12407ES |
| ชุดไพรเมอร์ Hieff NGS® Stubby UDI สำหรับ Illumina ชุดที่ 1-จาน 96 หลุม ดัชนี 1-96- ชุดที่ 1 | 12327ES | |
| ชุดไพรเมอร์ Hieff NGS® Stubby UDI สำหรับ Illumina ชุดที่ 2-จาน 96 หลุม ดัชนี 97-192- ชุดที่ 2 | 12328ES | |
| ชุดไพรเมอร์ Hieff NGS® Stubby UDI สำหรับ Illumina ชุดที่ 3-จาน 96 หลุม ดัชนี 193-288- ชุดที่ 3 | 12329ES | |
| ชุดไพรเมอร์ Hieff NGS® Stubby UDI สำหรับ Illumina ชุดที่ 4-จาน 96 หลุม ดัชนี 289-384- ชุดที่ 4 | 12330ES |
สำหรับ MGI
| ไฮฟ์ เอ็นจีเอส™ ชุดอะแดปเตอร์ UMI UDB คู่สำหรับ MGI ชุดที่ 1/ชุดที่ 2 | 96 ชนิดของดัชนี | |
| ไฮเอฟเอ็นจีเอส™ ชุดอะแดปเตอร์ครบชุดสำหรับ MGI ชุดที่ 1/ชุดที่ 2/ชุดที่ 3(สอบถาม) | 13360ES | 8 ประเภทดัชนี 41-48 |
| 13361ES | 16 ประเภทดัชนี 57-72 | |
| 13362ES | 96 ชนิดของดัชนี 1-96 |
เรื่องการอ่าน
เอนไซม์สำคัญที่เกี่ยวข้องกับการสร้างห้องสมุด NGS
คุณมีความรู้เกี่ยวกับเทคโนโลยีที่เกี่ยวข้องกับ NGS มากเพียงใด?
ลูกปัดแม่เหล็กชนิดต่างๆ ใน NGS: ลูกปัดแม่เหล็ก DNA\RNA\mRNA
การวัดปริมาณไลบรารี NGS: Qubit ที่รวดเร็วและแม่นยำหรือ qPCR ที่แม่นยำ จำเป็นทั้งหมด!
