| PCR -Master -Mix | Länge | Erweiterung | GC -Bereich | Farbstoff | Heißer Start | 3 ’End | Fidelity | Anwendung |
| ≤ 10-15 KB | 1-10 Sek./KB | 30-70% | Rose Rot | √ | 3'-da | 1 × | Genotypisierung, Kolonie PCR, schnelle PCR | |
| ≤ 5 KB | 30 Sek./KB | 40-70% | Blau | × | 3'-da | 1 × | reguläre PCR, Genotypisierung | |
| ≤5 kb | 30 Sek./KB | 40-70% | farblos | × | 3'-da | 1 × | reguläre PCR | |
| ≤4 kb | 30 Sek./KB | 30-70% | Blau | √ | 3'-da | 1 × | Mausgenotypisierung | |
| | | | | | | |
| |
| Direkte PCR | | | | | | | | |
| ≤ 10-15 KB | 1-10 Sek./KB | 30-70% | Rose Red | √ | 3'-da | 1 × | Colony Direct PCR | |
| ≤ 1 KB | 30 s/kb | 30-70% | Blau | √ | 3'-da | 1 × | Direkte PCR | |
| ≤8 kb | 10 s/kb für ≤8 kb | 30-75% | farblos | √ | 3'-da | 1 × | Blutdirekter PCR | |
| ≤ 1 kb | 60 s/kb | 40-65% | Blue | √ | 3'-da | 1 × | Direkte PCR | |
| | | | | | | | Mausgewebe/ Zelllyse -Reagenz | |
| | | | | | | | | |
| HOHE -FASHELITY PCR |
|
|
|
|
|
|
|
|
| ≤ 16 kb | 5 Sek./KB | 20-80% | blau | √ | Blunt | 83 × Taq | Vormix | |
| ≤13 KB | 30 Sek./KB | 30-60% | farblos | √ | Blunt | 83 × Taq | Enzym | |
| ≤13 kb | 30 Sek./KB | 30-60% | Blau | √ | Blunt | 83 × Taq | Vormischung | |
| | | | | | | | | |
| Klonierungskit | Nr.der Fragmente Ligation | Fragmentlänge | Volumen | Vormix | DSDNA -Assemblierung | ssDNA -Assemblierung |
| Name |
| 1-6 | 50 bp-25 kb | 20 & mgr; l oder 6 & mgr; l | Ja | √ | × |
| Hieff -Klon -Universal II Ein Schritt Klonierungskit |
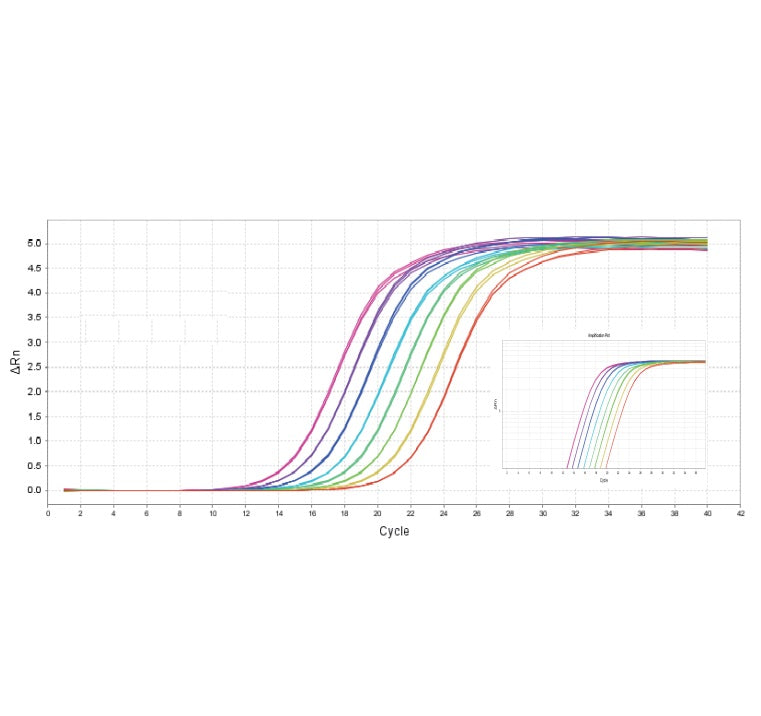
Reverse-Transkription und Echtzeit-QPCR-Master-Mix
| qpcr | Nachweis | rox | Empfindlichkeit | Spezifität | High GC | Komponenten |
| Farbstoffbasierte | NO/LOW/HIGH ROX | 9 | 9 | 9 | blau qpcr mix | |
| Farbstoffbasierte | no rox | 9 | 9 | 8 | blau qpcr mix + gelbe template diluent | |
| Farbstoffbasierte | Low Rox | 9 | 9 | 8 | Blau QPCR Mix + Gelbe Template Diluent | |
| Dye-basierte | High Rox | 9 | 9 | 8 | Blau QPCR Mix + Gelbe Template Diluent | |
| Farbstoffbasierte | NO/LOW/HIGH ROX | 9 | 9 | 9 | miRNA qPCR + H2O | |
| Sondenbasierte | NO/LOW/High Rox | 10 | 9 | 8 | Master -Mix | |
| | 50 × Rox Referenzfarbstoff | | | | Referenzfarbstoff | |
|
|
|
|
|
|
|
|
| RT-qPCR | Produktpositionierung | Multiplex qPCR | Empfindlichkeit | Spezifität | High GC | kompatible Plattformen |
| Farbstoffbasierte |
| 9 | 9 | 8 | NO/LOW/HIGH ROX | |
| Basis basierend | √ | 10 | 9 | 8 | NO/LOW/HIGH ROX | |
|
|
|
|
|
|
|
|
| Reverse -Transkription | RT -Temperatur | Hoch -GC oder komplexe Vorlage | RT -Zeit | GDNA -Entferner | Für qPCR | Sub-Application |
| 55℃ | High | 15 min | √ | √ |
| |
| 37℃ | Gut | 50 min | × | √ | miRNA | |
| 50℃ | High | 5 min | √ | √ |
| |
| 37℃ | GUT | 5 min | √ | √ |
| |
| 55℃ | High | 5 min | √ | √ | PCR, ≤ 14 KB |
Nukleinsäureelektrophorese
| Kategorie | CAT. No. | Produktname | Anwendung |
| agarose | Agarose | Routinemerzer -Nukleinsäurelektrophorese | |
| Hohe Sieb -Agarose (PCR -Grad) | Geeignet zur Trennung von DNA-Fragmenten von 20 bp-800 bp, vergleichbar mit Polyacrylamidgel | ||
| Agarose -Tabletten (0.5 g/tablet) | Bequem für routinemäßige Agaroseanwendungen | ||
| Nukleinsäure -Farbstoff | Jahresnukleinsäure -Gel -Färbung (10.000 × in Wasser) | wasserlöslich mit spektralen Eigenschaften, die EB ähneln, erregt bei 300 nm UV-Licht | |
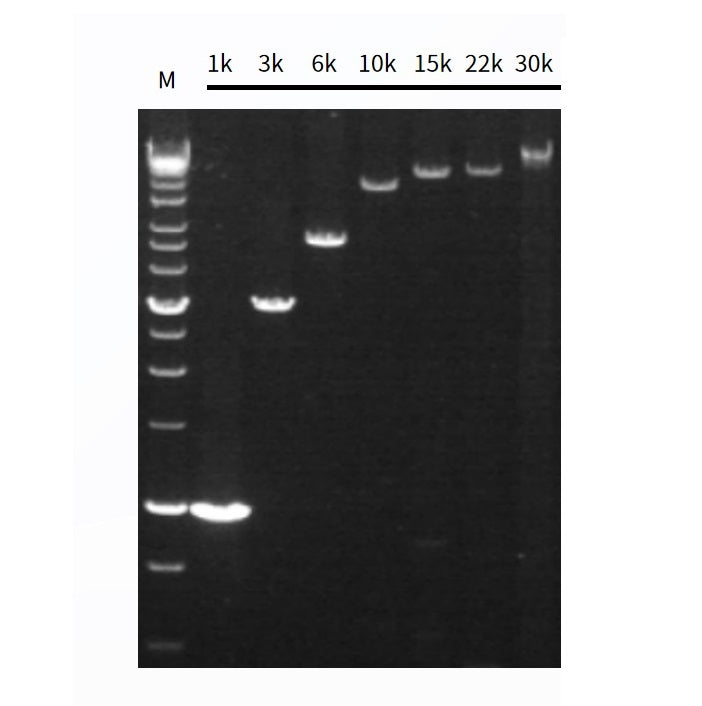
| DNA -Marker | Goldband 1 kb DNA -Leiter | 250-12000 bp (13 Bänder) | |
| Goldband 50 bp DNA -Leiter | 50-1000 bp (14 Bänder) | ||
| Goldband 100BP -DNA -Leiter | 100-1500 bp (12 Bänder) | ||
| Goldband 100 bp plus DNA -Leiter | 100-3000 bp (14 Bänder) | ||
| Goldband 200 bp DNA -Leiter | 200-5000 bp (12 Bänder) | ||
| Goldband 500 bp DNA -Leiter | 500-5000 bp (8 Bänder) | ||
| Goldband DL2000 DNA -Marker | 100-2000 bp (6 Bänder) | ||
| Goldband DL5000 DNA -Marker | 100-5000 bp (9 Bänder) | ||
| Goldband DL10.000 DNA -Marker | 100-10000 bp (10 Bänder) | ||
| DNA-Leiter der Goldband-DNA | 100-12000 bp (20 Bänder) | ||
| Goldband DL15000 DNA -Marker | 250-15000 bp (7 Bänder) |