Lavora su entrambe le linee, analisi dell'espressione miRNA semplificata
La lunghezza del miRNA maturo è di soli 20nt circa, di solito il primer forward coprirà la sua lunghezza completa o avrà anche una parte in eccesso, e il primer reverse non avrà alcun posto dove essere posizionato. L'attuale soluzione di mercato è quella di aumentare la lunghezza della trascrizione inversa durante la trascrizione inversa. I metodi comuni includono il metodo tailing e il metodo stem-loop.
1. Introduzione
2. Prodotti consigliati
3. Elenco prodotti correlati
1. Introduzione
I microRNA (miRNA) sono piccoli RNA a singolo filamento con una dimensione di circa 21-23 basi, che vengono generati da precursori di RNA a singolo filamento con una struttura a forcina di circa 70-90 basi dopo l'elaborazione enzimatica Dicer. I miRNA si legano principalmente in modo specifico alla regione non codificante all'estremità 3' dell'mRNA del gene bersaglio e regolano il processo di traduzione dell'mRNA del gene bersaglio post-trascrizionale, bloccando così la traduzione dell'mRNA e promuovendo la degradazione dell'mRNA, con conseguente riduzione dell'espressione proteica. Il ruolo dei miRNA è coinvolto nell'insorgenza e nello sviluppo di varie malattie cardiovascolari come l'ontogenesi, la differenziazione tissutale, la proliferazione cellulare e l'apoptosi.
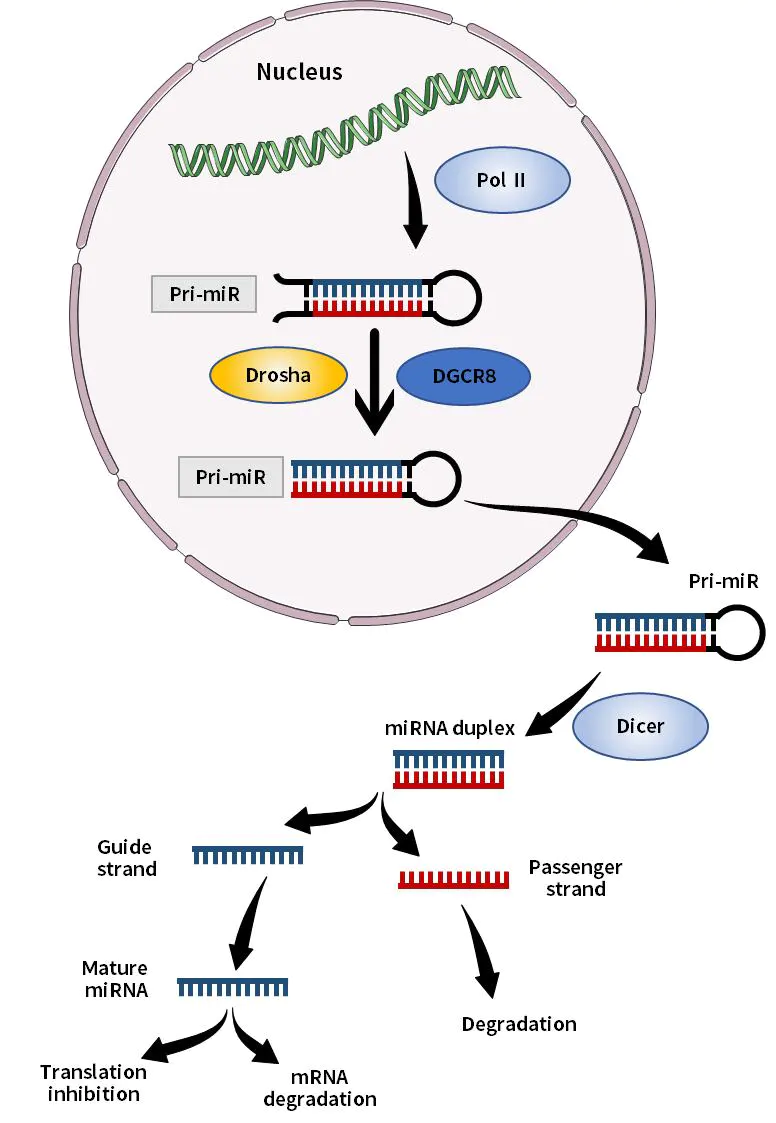
Figura 1. Diagramma schematico della sintesi di miRNA in vivo
La lunghezza del miRNA maturo è di soli 20nt circa, di solito il primer forward coprirà la sua lunghezza completa o avrà anche una parte in eccesso, e il primer reverse non avrà alcun posto dove essere posizionato. L'attuale soluzione di mercato è quella di aumentare la lunghezza della trascrizione inversa durante la trascrizione inversa. I metodi comuni includono il metodo tailing e il metodo stem-loop.
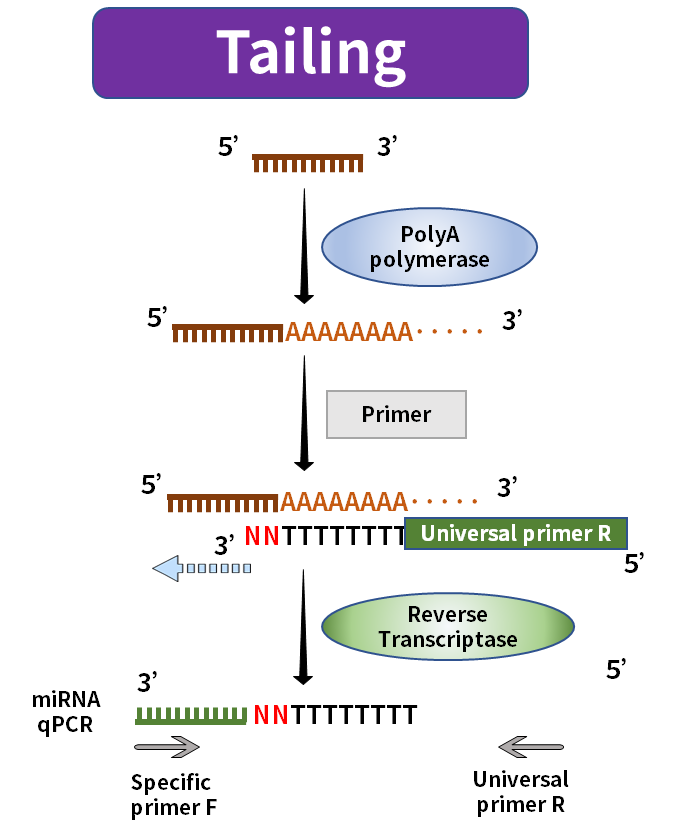
Figura 2. Sintesi di miRNA mediante il metodo tailing
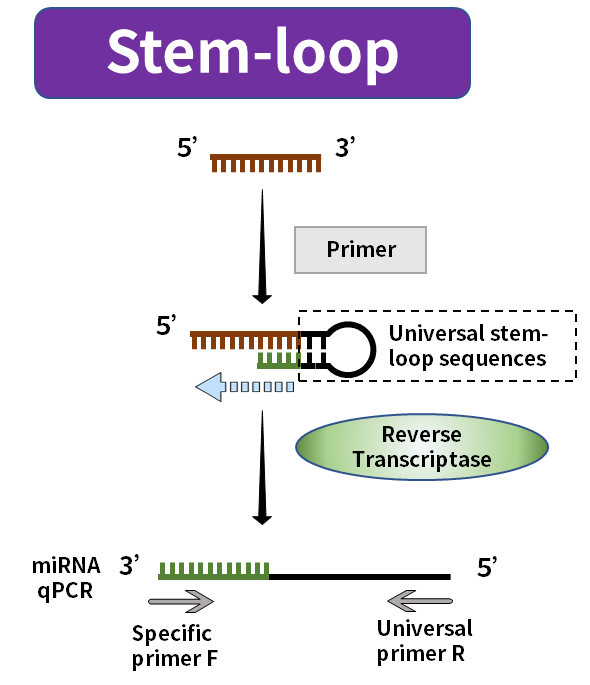
Figura 3. Sintesi di miRNA con il metodo stem-loop
Il metodo di tailing utilizza la polimerasi PolyA per aggiungere la coda Poly(A) al miRNA maturo e utilizza Oligo-dT con la sequenza universale come primer di trascrizione inversa per sintetizzare il miRNA allungato corrispondente al primo filamento di cDNA. Il metodo stem-loop utilizza la sequenza universale ripiegata nella struttura stem-loop come primer per sintetizzare il primo filamento di cDNA corrispondente al miRNA allungato. La struttura stem-loop verrà aperta a una temperatura adatta durante il successivo processo di amplificazione e combinati con i primer di amplificazione pertinenti.
Tabella 1.Confronto tra Tailing e Stem-loop
| Categoria | Coda | Stelo-anello |
| Applicabilità | Adatto per studiare simultaneamente più miRNA diversi | Adatto per il rilevamento quantitativo accurato di alcuni miRNA in un campione |
| Vantaggio | Una singola trascrizione inversa può rilevare più miRNA con elevata produttività | Solo un miRNA può essere rilevato tramite trascrizione inversa alla volta, con forte specificità |
| Sistema | Insieme al miRNA coinvolto, la trascrizione inversa nello stesso sistema di reazione con elevata accuratezza | I miRNA coinvolti internamente vengono separati e trascritti inversamente nei due sistemi, il che è soggetto a errori |
| Specificità | ★★★★☆ | ★★★★★ |
| Sensibilità | ★★★★★ | ★★★★☆ |
| Tempo | ★★★★☆ | ★★★★★ |
2. Prodotti consigliati
2.1 Combinazione quantitativa di inversione della coda di miRNA Yeasen (11148ES&11171ES)
Figura 4. Kit di sintesi del cDNA del 1° filamento di miRNA 11148ES-Hifair™
Il kit adotta il metodo di tailing per la trascrizione inversa del cDNA del primo filamento di miRNA. Il tampone RT miRNA 2×Hifair™ nel prodotto contiene tutte le materie prime e i primer per la reazione di tailing miRNA e la reazione di trascrizione inversa, ed è stato attentamente ottimizzato. Può garantire che i processi di modifica del poli(A) e di trascrizione inversa dell'estremità 3' del miRNA vengano eseguiti in modo efficiente simultaneamente.
Figura 5. 11171ES -Hieff™ miRNA Universal qPCR SYBR Master Mix
Una nuova generazione di reagenti di rilevamento PCR quantitativo a fluorescenza premiscelati, sviluppati appositamente per il rilevamento quantitativo di miRNA, contenenti uno speciale colorante di riferimento passivo ROX, adatti a tutti gli strumenti qPCR, non è necessario regolare la concentrazione di ROX su diversi strumenti, solo nella reazione di preparazione. L'amplificazione può essere eseguita aggiungendo primer e modelli al sistema. La DNA polimerasi adotta una polimerasi hot-start modificata chimicamente e collabora con uno speciale sistema di buffer, che aumenta la specificità e la sensibilità della reazione e può quantificare accuratamente in un intervallo più ampio.
2.2 Prestazioni del prodotto
2.2.1 Compatibile con tutti gli strumenti qPCR della piattaforma

Figura 6. Risultati compatibili con diversi strumenti qPCR
Utilizzando l'RNA totale delle cellule 293T come modello, è stata eseguita la trascrizione inversa con il kit di sintesi del cDNA a 1 filamento Hifair™ miRNA (Cat# 11148ES).Il cDNA ottenuto è stato diluito 10 volte e poi utilizzato Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) per rilevare rispettivamente i geni di riferimento interni di hsa-miR-let-7b-5p, hsa-miR-let-7c-5p e U6.
2.2.2 Elevato tasso di rilevamento, fino a 10 pg
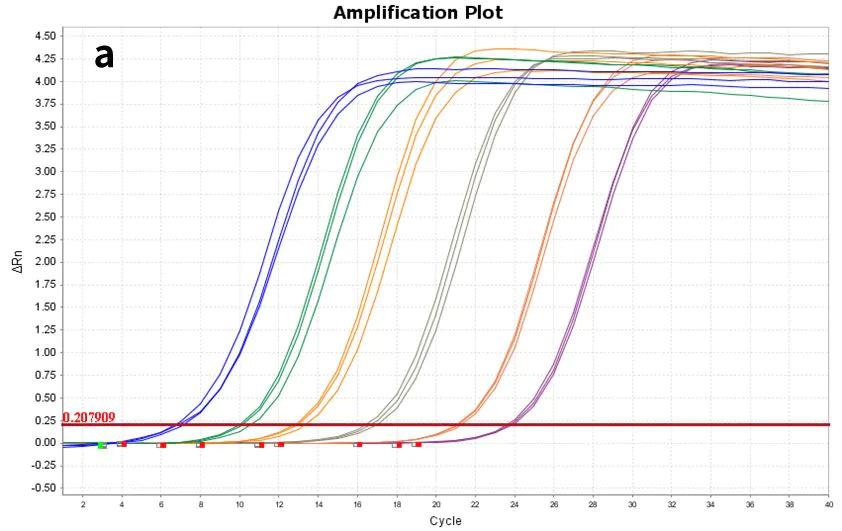
Figura 7. Grafico di amplificazione
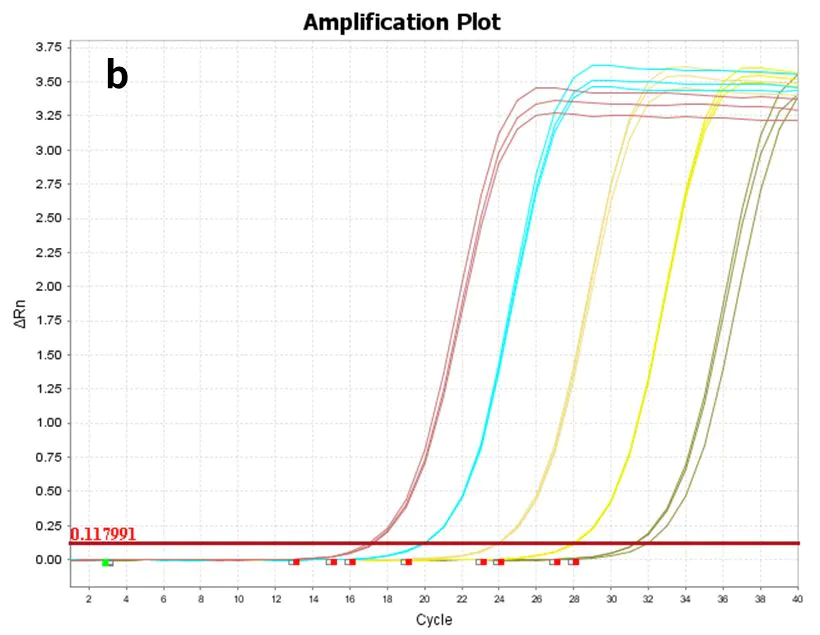
Figura 8. Grafico di amplificazione
Utilizzando hsa-miR-let-7e-5p(a) sintetico e RNA(b) totale delle cellule 293T come modelli, diluiti rispettivamente nei seguenti gradienti: da 60 copie a 606 copie (6 gradienti) e da 10 pg a 100 ng (5 gradienti), sottoposti a trascrizione inversa con il kit di sintesi cDNA a 1 filamento Hifair™ miRNA (Cat# 11148ES), e il cDNA risultante è stato rilevato con Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) per l'espressione genica hsa-miR-let-7e -5p.
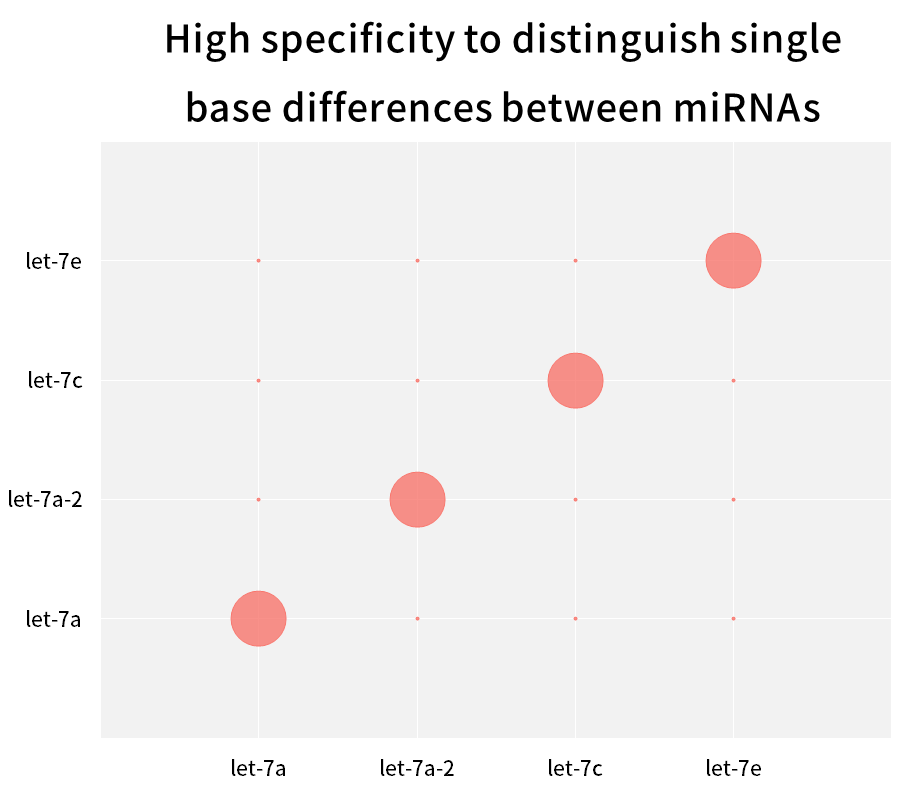
2.2.3 Elevata specificità, in grado di distinguere le differenze di singole basi tra miRNA della stessa famiglia
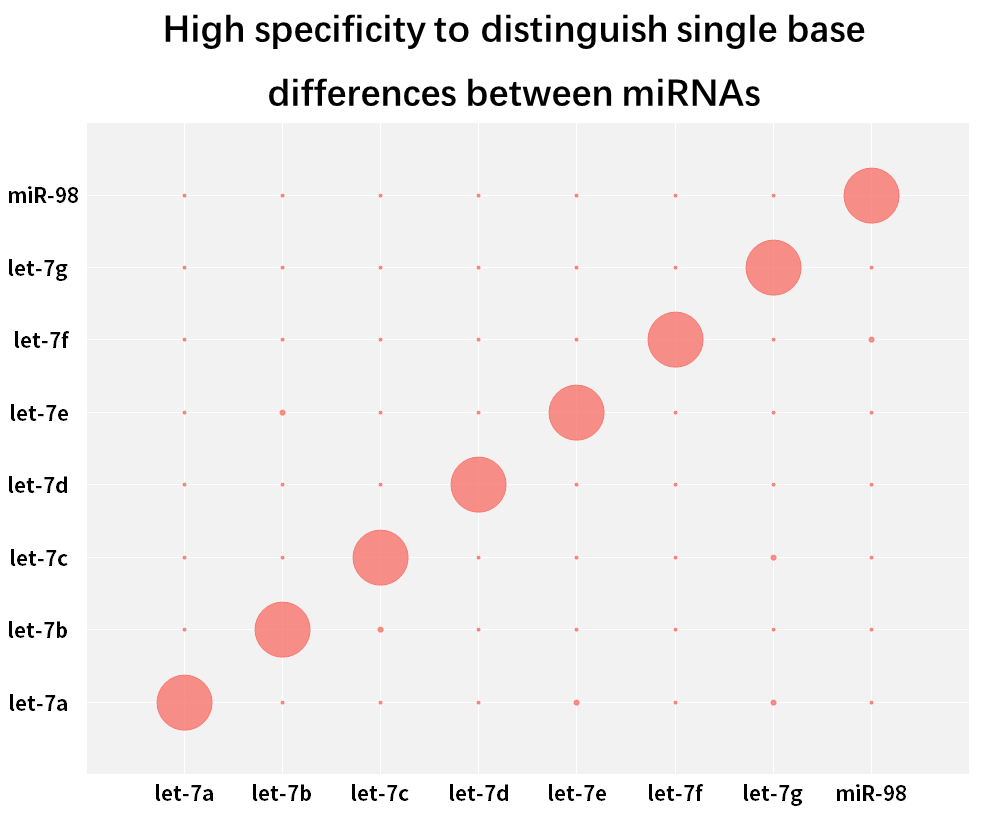
Figura 9. Distinguere le differenze di singole basi tra i miRNA
La famiglia umana hsa-let-7 ha più miRNA, con poche basi che differiscono tra loro, o anche solo una differenza di una base. I miRNA sintetici per ogni isoforma Let-7 (hsa-miR-let-7a~Let-7i, has-miR-98) sono stati trascritti inversamente in cDNA utilizzando l'Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat# 11148ES), utilizzando Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) per amplificare questi miRNA separatamente con diversi primer e calcolare il tasso di corrispondenza utilizzando l'equazione 2-ΔCT x 100%. I risultati mostrano che i membri della famiglia di sottotipi strettamente correlati possono essere distinti in modo efficace.
2.2.4 Ottima efficienza di amplificazione
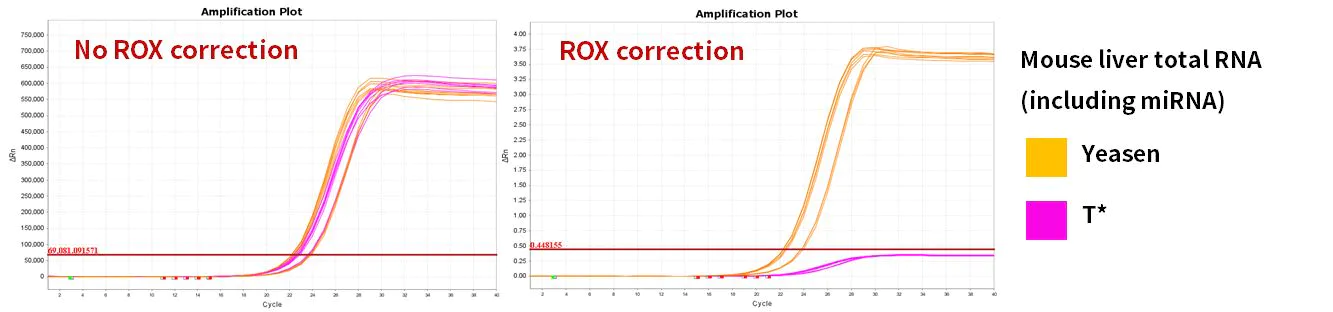
Figura 10. Efficienza di amplificazione
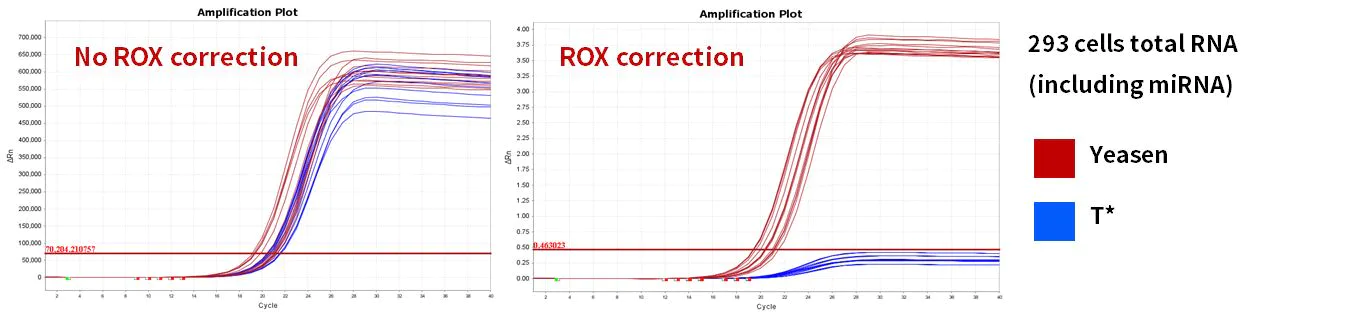
Figura 11. Efficienza di amplificazione
I geni miR-let-7b-5p, miR-let-7c-5p, miR-let-7e-5p, miR-let-7f-5p sono stati amplificati utilizzando cellule 293T umane e RNA totale di fegato di topo come modelli. I risultati hanno mostrato che, rispetto a prodotti simili, Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Cat# 11148ES) ha abbinato il sistema di reazione di Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES) con prestazioni eccellenti.
2.2.5 Ottima stabilità

Figura 12. Analisi della stabilità del prodotto a 37℃ per 7 giorni

Figura 13. Analisi dei risultati dopo 50 cicli di congelamento-scongelamento
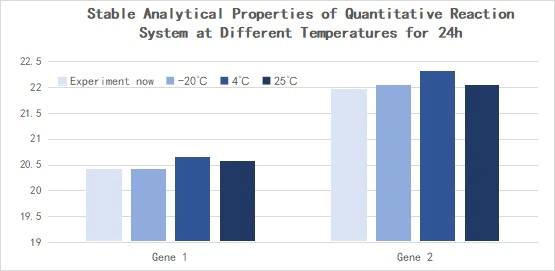
Figura 14. Proprietà analitiche stabili del sistema di reazione quantitativa a diverse temperature per 24 ore
La trascrizione inversa è stata eseguita con il kit di sintesi cDNA 1st Strand miRNA Hifair™ (Cat# 11148ES) utilizzando l'RNA totale delle cellule 293T come stampo e il cDNA ottenuto è stato diluito 10 volte e rilevato con Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11171ES). Espressione di hsa-miR-let-7b-5p, hsa-miR-let-7c-5p, hsa-miR-let-7e-5p. △Ct<0,5.
2.3 Combinazione quantitativa di inversione del ciclo stelo-ansa del miRNA Yeasen (11139ES&11170ES)
Figura 15. Kit di sintesi del cDNA a 1° filamento 11139ES-Hifair™ III (digestore gDNA più)
Il kit è stato sviluppato sulla base della trascrittasi inversa Hifair™ III.La velocità di sintesi del cDNA enzimatico è elevata e la stabilità termica è notevolmente migliorata. Allo stesso tempo, l'enzima aumenta l'affinità con il modello ed è adatto per la trascrizione inversa di una piccola quantità di modelli e geni a bassa copia. È stata inoltre migliorata la capacità della trascrittasi inversa Hifair™ III di sintetizzare cDNA a lunghezza intera, consentendo la sintesi di cDNA fino a 19,8 kb.

Figura 16. 11170ES-Hieff™ miRNA Universal qPCR SYBR Master Mix
Questo prodotto è uno speciale master mix per la quantificazione dei miRNA utilizzando il metodo di fluorescenza chimerica SYBR Green I. Poiché le sequenze dei miRNA sono corte e le sequenze dei miRNA nella stessa famiglia sono spesso molto simili, la specificità è estremamente richiesta durante la quantificazione. Questo prodotto si basa sulla DNA polimerasi hot-start, con tampone ottimizzato, che può ridurre notevolmente l'amplificazione non specifica; allo stesso tempo, lo speciale colorante di riferimento ROX rende il master mix adatto a tutti gli strumenti qPCR e non è necessario regolare ROX su strumenti diversi. L'amplificazione può essere eseguita aggiungendo primer e modelli durante la preparazione del sistema di reazione.
2.3.1 Compatibile con tutti gli strumenti qPCR della piattaforma
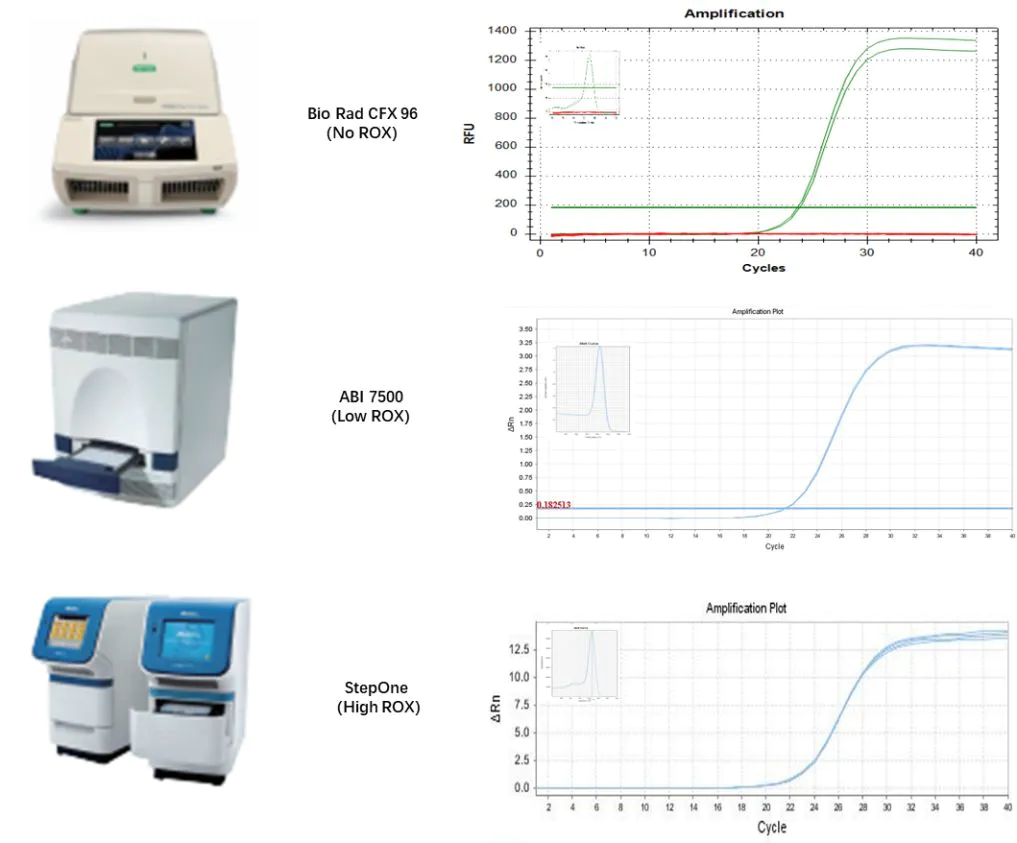
Figura 17. Risultati compatibili con diversi strumenti qPCR
Utilizzando l'RNA totale delle cellule epatiche del topo come modello, è stata eseguita la trascrizione inversa con Hifair™ III 1st Strand
Kit di sintesi del cDNA (gDNA digester plus) (Cat# 11139ES), e il cDNA ottenuto è stato diluito 40 volte e utilizzato Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES) per amplificare rispettivamente il gene umano mmu-miR-125a.
2.3.2 Alta sensibilità, fino a 10 pg

Figura 18. Rilevamento della sensibilità
Utilizzando l'RNA totale delle cellule epatiche del topo come modello, è stata eseguita la trascrizione inversa con il kit di sintesi del cDNA a 1 filamento Hifair™ III (gDNA digester plus) (codice articolo 11139ES) e il cDNA ottenuto è stato diluito in serie 10 volte (intervallo 10pg/μL). -0,1 μg/μL), amplificano i geni mmu-miR-125a, mmu-miR-132 e mmu-miR-351 di origine murina.
2.3.3 Elevata specificità, in grado di distinguere le differenze di singole basi tra miRNA della stessa famiglia
Figura 19. Distinguere le differenze di singole basi tra i miRNA
La famiglia umana hsa-let-7 ha più miRNA, con poche basi che differiscono tra loro, o anche solo una differenza di una singola base. Questi miRNA sono stati amplificati separatamente con diversi primer usando Hieff™ miRNA Universal qPCR SYBR Master Mix (Cat# 11170ES), e il tasso di corrispondenza è stato calcolato usando la formula 2^-Δct x 100%.
2.3.4 Buona ripetibilità

Figura 20. Rilevamento della ripetibilità
Utilizzando l'RNA totale delle cellule umane 293T come modello per la trascrizione inversa, la trascrizione inversa è stata eseguita con il kit di sintesi cDNA a 1 filamento Hifair™ III (gDNA digester plus) (n. cat. 11139ES) e il cDNA ottenuto è stato diluito 50 volte e utilizzato. Hieff™ miRNA Universal qPCR SYBR Master Mix (n. cat. 11170ES) è stato utilizzato per amplificare il gene umano hsa-let-7a in un esperimento replicato a 90 pozzetti.
2.3.5 Buona stabilità

Figura 21.Analisi della stabilità di 11170ES a diverse temperature per 14 giorni
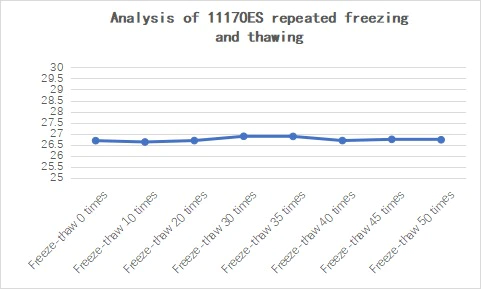
Figura 22. Analisi del congelamento e scongelamento ripetuti di 11170ES

Figura 23. Proprietà di analisi stabili del sistema di reazione quantitativa a diverse temperature per 24 ore
L'RNA totale delle cellule epatiche del topo è stato utilizzato come modello per la trascrizione inversa con il kit di sintesi cDNA 1st Strand Hifair™ III (gDNA digester plus) (Cat# 11139ES). Il cDNA ottenuto è stato diluito 100 volte per amplificare il gene murino mmu-miR-132.
3. Elenco prodotti correlati
Yeasen Biotechnology (Shanghai) Co., Ltd., fondata nel 2014, è un'impresa high-tech impegnata nella R&S e nella produzione di materie prime enzimatiche per utensili e anticorpi antigeni. I suoi prodotti includono enzimi diagnostici molecolari, proteine e anticorpi utilizzati in prodotti farmaceutici, test di sicurezza alimentare, allevamento, giustizia e altri settori. Ci impegniamo a fornire ai clienti nel campo delle scienze della vita prodotti e servizi di alta qualità. I prodotti correlati forniti da Yeasen sono i seguenti:
Tabella 2. Elenco dei prodotti correlati
| Tipo di metodo | Procedura sperimentale | Nome del prodotto | Codice prodotto |
| Coda | Trascrizione inversa | Alta qualità™ Kit di sintesi del cDNA del 1° filamento di miRNA | 11148ES |
| PCR-q | 11171ES | ||
| Stelo-anello | Trascrizione inversa | Alta qualità™ III Kit di sintesi del cDNA a 1° filamento (digestore gDNA plus) (Richiesta di informazioni) | 11139ES |
| PCR-q | Materiale miRNA Universal qPCR SYBR Master Mix (Richiesta di informazioni) | 11170ES |